qPCR in der Abwasseranalytik: Von der Herausforderung zur Innovation
Warum die qPCR-Technologie bisher noch nicht ihren Weg in die Abwasserwelt fand...
In der Vergangenheit wurde vor allem die Mikroskopie verwendet, um Mikroorganismen im Abwasser zu bestimmen und zu quantifizieren weil das Mikroskop eine klare Unterscheidung zwischen lebenden und abgestorbenen Zellen ermöglichte. Da für Kläranlagenbetreiber nur die lebenden und aktiven Mikroorganismen für die Reinigungsleistung relevant sind, war die Mikroskopie hierfür besonders geeignet.
Heute hat sich die Real-Time qPCR-Technologie in verschiedensten Bereichen wie der klinischen Diagnostik oder Lebensmittelanalytik als die Methode der Wahl etabliert, wenn schnell, hochspezifisch und zuverlässige Ergebnisse benötigt werden. Diese Technologie kann normalerweise jedoch nicht zwischen Genfragmenten unterscheiden, die von lebenden oder bereits abgestorbenen Mikroorganismen stammen. Dies war ein bedeutender Nachteil der qPCR-Methode und der Hauptgrund, warum sie in der mikrobiologischen Abwasseranalytik bisher nur begrenzte Anwendung fand.
Von der Herausforderung mit der qPCR-Technologie nur lebendige Bakterien im Abwasser zu detektierten...
Die DyeNA Genetics hat ein innovatives Verfahren entwickelt mit dem die qPCR so gesteuert wird, dass nur Genfragmente aus einer Abwasserprobe erkannt werden, die von lebendigen und somit aktiven Bakterien stammen.
Dies geschieht mit einer Methode bei der die Gene aus toten Bakterien für die qPCR nicht zugänglich gemacht werden. Hierdurch werden tote oder geschädigte Bakterienzellen aus der qPCR-Quantifizierung effektiv ausgeschlossen.
Durch diese Innovation wird der frühere Nachteil der qPCR, nämlich die fehlende Unterscheidung zwischen lebenden und abgestorbenen Bakterien, aufgehoben. Nun können auch Kläranlagen von dieser präzisen, hochspezifischen und zuverlässigen Technologie profitieren, die eine genaue Bestimmung der bakteriellen Lebendzellzahl im Abwasser ermöglicht, und das mit einer höheren Aussagekraft als bei der Mikroskopie.
Wie man mit der qPCR-Methode ein einzelnes Bakterium oder Virus in der Probe nachweisen kann...
Die quantitative Polymerase-Kettenreaktion (qPCR) ist eine leistungsstarke Technik aus der Molekularbiologie. Mit dieser Technik ist es ermöglicht, selbst einzelne Gensequenzen, die von einem einzelnen Bakterium oder Virus stammen, gezielt zu vervielfältigen und hierdurch zu quantifizieren.
Das Grundprinzip der qPCR besteht darin, dass ein bakterienspezifischer Genabschnitt in der Probe milliardenfach vervielfältigt wird. Dies geschieht in einem speziellen Gerät, dem Thermocycler. Dabei werden die DNA-Stränge der Gene zuerst auf hohe Temperaturen erhitzt, um sie zu trennen. Anschließend kühlt das Gerät die Probe ab, damit speziell entwickelte kurze DNA-Abschnitte, die sogenannten Primer und die Fluoreszenz-Gensonden, an die DNA binden können.
Das Enzym Taq-Polymerase fügt dann neue DNA-Bausteine an die DNA-Stränge an, wodurch die Anzahl der DNA-Moleküle exponentiell vervielfältigt wird. Während dieses Vorgangs wird die am Zielgen gebundene Fluoreszenz-Gensonde abgebaut und der Fluoreszenzfarbstoff freigesetzt. Dieser beginnt zu leuchten und hierdurch kann die Genmenge in einer Probe präzise quantifiziert werden. Durch diese Vervielfältigung der Gene ist es möglich selbst ein einziges Bakterium oder Virus in einer Probe zu detektieren. Dies verleiht der qPCR-Technik eine sehr hohe Nachweisgrenze.
Durchführung einer Real-Time qPCR Analyse
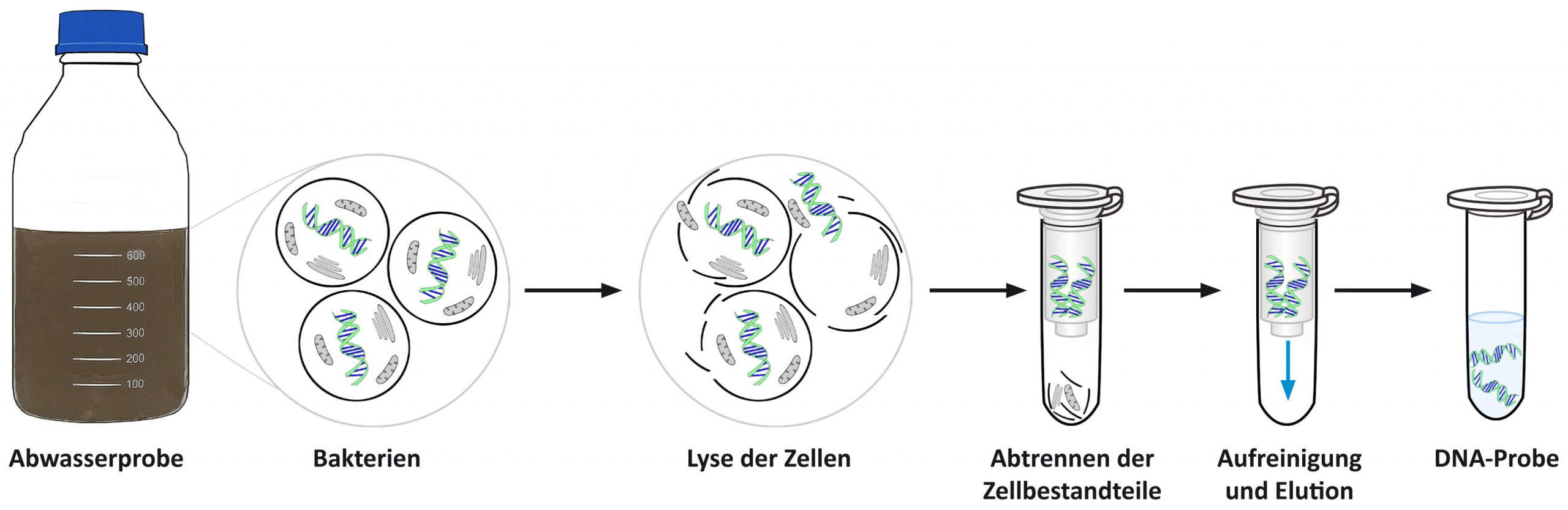
Schritt 1: Extraktion von DNA direkt aus der Abwasserprobe
Zunächst werden die Zellwände der Mikroorganismen in der Probe aufgebrochen, um die DNA-Moleküle freizusetzen. Dies wird mittels eines speziellen Verfahrens, der DNA-Extraktion, erreicht. Die resultierende Lösung, die sowohl freie DNA als auch aufgebrochene Zellfragmente enthält, wird auf Extraktionssäulen aufgetragen. Diese Säulen ermöglichen es, die Zellfragmente von der freien DNA zu trennen. Die DNA heftet sich an die Säulenmatrix und wird zurückgehalten, während die Zellfragmente die Säule passieren. Zum Schluss wird die DNA mithilfe eines speziellen Puffers von der Säule gelöst. Die extrahierte DNA ist von hoher Reinheit und spiegelt die ursprüngliche mikrobiologische Situation in der Probe genau wider. Diese extrahierte DNA dient als Ausgangsmaterial für die anschließende qPCR-Analyse.
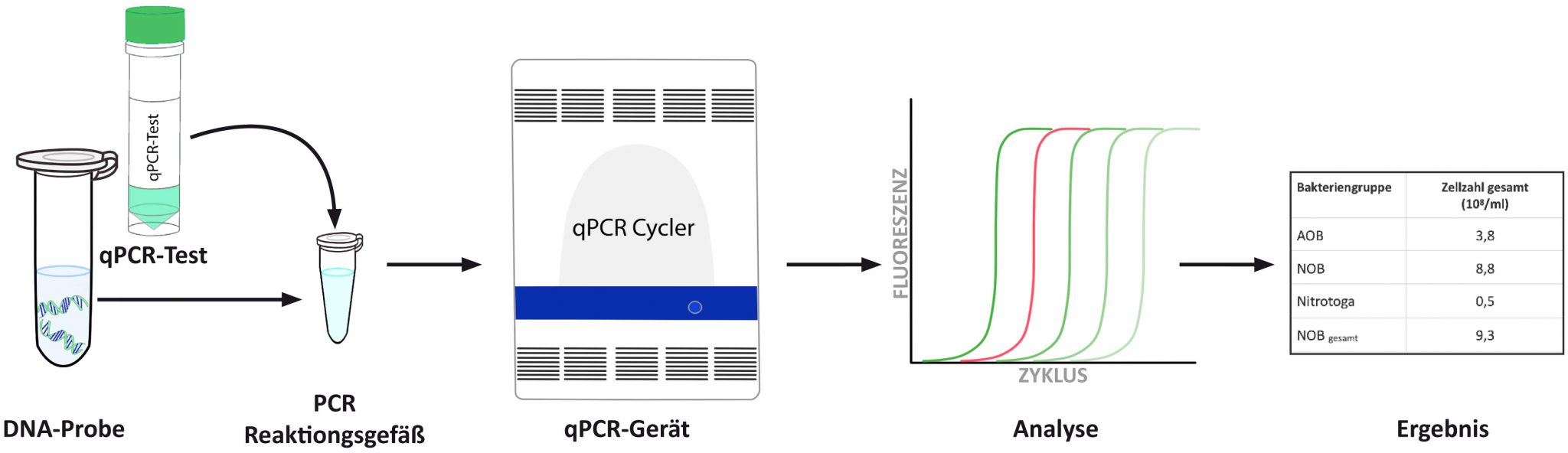
Schritt 2: qPCR-Analyse
Die zuvor extrahierte DNA-Probe wird mit einem bakterienspezifischen qPCR-Mix gemischt, in ein PCR-Reaktionsgefäß pipettiert und in den Thermocycler platziert. Durch zyklisches Erhöhen bzw. Absenken der Temperatur im Reaktionsgefäß werden die DNA-Moleküle enzymatisch vervielfältigt. Dabei entsteht das Fluoreszenzsignal. Am Ende der qPCR-Analyse berechnet eine Software anhand der Fluoreszenzintensität im Reaktionsgefäß die Menge der DNA in der ursprünglichen Probe. Diese computergestützte Auswertung verhindert Fehler und Ungenauigkeiten, die durch menschliche Interpretation entstehen könnten. Die gesamte Analysezeit für 96 Proben beträgt etwa 40-45 Minuten.
Das Basis- und Flex-System – Analysen maßgeschneidert an Ihre Anforderungen
Das extrahierte DNA-Volumen ermöglicht die simultane Identifizierung von bis zu 8 Bakterientypen aus einer Probe. Da die DNA-Isolation nur einmal pro Probe erfolgt, können zusätzliche qPCR-Analysen aus derselben Probe durchgeführt und kostengünstiger angeboten werden.
Basis-Analysen umfassen die DNA-Extraktion und entsprechende qPCR-Tests.
Flex-Analysen nutzen die zuvor isolierte DNA aus Basisanalysen für zusätzliche qPCR-Tests. Die Auswahl der zusätzlichen Analysen ist variabel. Flex-Analysen sind nur in Kombination mit Basis-Analysen möglich.
Ihre Vorteile von Basis- und Flex-Analysen
- Die DNA-Extraktion wird nur einmal berechnet, es entstehen keine unnötigen Kosten
- Dienstleistungen ideal angepasst an Ihre Anforderungen
- Multiparameter-Bestimmungen aus ein und derselben Probe
- Bis zu 8 Bestimmungen gleichzeitig aus einer Probe
- Flex-Analysen können auf jeden Probentyp angewendet werden
- Unterschiedliche qPCR-Bestimmungen sind nach Bedarf frei kombinierbar
- Kostenreduktion durch maßgeschneiderte Analysendientleistungen
Häufig gestellte Fragen:
Mit unseren Tests können eine Vielzahl von Wasserproben (Zulauf, Belebungsbecken, Ablauf, Prozesswasser und Faulturm einer Kläranlage, Flusswasser, Brunnenwasser und Brauchwasser) untersucht werden.
Die Handhabung hierfür ist denkbar einfach. Die DyeNA Genetics schickt Ihnen die Probenröhrchen und bereits frankierte Briefumschläge zu. Die Probe wird in das Röhrchen gefüllt, in den Briefumschlag gegeben und mit der Post verschickt.
In der Regel trifft die Probe mit der Post am nächsten Tag in unserem Labor ein. Das Analysenergebnis wird normalerweise binnen 48 Stunden nach Probeneingang elektronisch zugestellt.
Die Abkürzung VBNC steht im Englischen für „viable but non-culturable“, was so viel bedeutet wie „lebensfähig, aber nicht kultivierbar“ in Bezug auf Bakterien.
Basierend auf heutigem Wissen können nur wenige Bakterien aus Umweltproben unter Laborbedingungen kultiviert werden. Der Großteil bleibt mit künstlichen Nährmedien unentdeckt. Hinzu kommt eine Gruppe von Bakterien, die grundsätzlich nicht kultiviert werden können.
Mithilfe der qPCR-Technologie können Bakterien direkt in der Probe ohne Notwendigkeit der Kultivierung nachgewiesen werden. Dadurch wird ein umfassendes und direktes Bild der vorhandenen Bakterienarten in der Probe vermittelt.
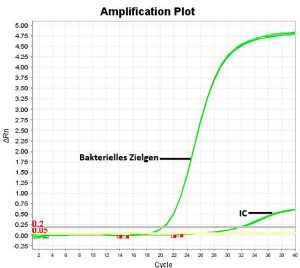
In der medizinischen Diagnostik kommen hauptsächlich Duplex-Testsysteme zum Einsatz. Dabei werden sowohl das bakterielle Zielgen als auch eine „interne Kontrolle“ (IC) gleichzeitig erfasst (siehe Abbildung rechts).
Die IC ist ein üblicherweise synthetisches DNA- oder RNA-Fragment, das als Kontrolle dem Abwasserprobe beigefügt wird. Die IC unterliegt dem gesamten Reinigungsprozess und wird schließlich zusammen mit der extrahierten DNA oder RNA von der Säule eluiert.
In der qPCR-Analyse wird die IC quantifiziert und ihr Wert muss innerhalb eines bestimmten Bereichs liegen.
Dies gewährleistet, dass Probleme wie Enzymreaktionshemmung oder Störungen bei der RNA/DNA-Extraktion sofort erkannt werden.Die Verwendung eines Duplex qPCR-Ansatzes sichert die Zuverlässigkeit unserer Testsysteme ab, gewährleistet verlässliche Ergebnisse und trägt zu einer hohen Qualität unserer Analysen bei.
Bei Multiplex-Tests werden mehrere Bakterienarten in einer qPCR-Analyse gleichzeitig in der gleichen Probe nachgewiesen. Zum Beispiel bei unserer qPCR-Bestimmung der Nitrifikanten werden die Amonium- bzw. die Nitrit-oxidierenden Bakterien und Nitrotoga aus der gleichen DNA-Probe bestimmt.
Die gleichzeitige Analyse liefert sehr zuverlässige Ergebnisse da mögliche Schwankungen innerhalb des Probenmaterials augeschlossen werden. Alle unsere Multiplextest sind ebenfalls durch eine interne Kontrolle (s. Duplex-Tests) abgesichert, die unseren Messergebnissen eine hohe Analysenqualität verleiht.
Die fehlende Fähigkeit der Lebendzellzahlbestimmung war der Grund weshalb die qPCR-Methode in der Vergangenheit suboptimal für den Einsatz in der Abwasseranalytik war und andere Techniken wie z.B. die Mikroskopie den Vorzug erhielten.
Die Testsysteme der DyeNA Genetics sind in der Lage die Lebendzellzahl mittels qPCR-Methode zu bestimmen.
Somit vereinen unsere Tests die Vorteile der qPCR Technologie wie Schnelligkeit, Genauigkeit und Präzsion mit dem Vorteil der Lebendzellzahlbestimmung aus der Mikroskopie zu neuen, innovativen Tests für die Abwasseranalytik.
